データドリブン・ライフサイエンス
新しい生物学に挑戦している久米さんからのご紹介で、京田耕司さんにネホリハホリ聞いてきました。今回は、長くて難しいかも……。でも、ここ最近のAIやビッグデータの流れとライフサイエンスを考える上でとても勉強になりましたので、最後までお付き合いいただければ幸いです。(聞き手:薬師寺秀樹)
データからネットワーク推定
京田さんといえば、大浪チームの大番頭なイメージがありますね。
問題児ですね(苦笑)。付き合いは僕が修士の頃からなので、かなり長いです。そのとき僕自身は北野ERATO北野共生システムプロジェクト。https://www.jst.go.jp/erato/research_area/completed/kks2_PJ.html
システムズバイオロジーの発展に大きく貢献した。にいたんですけどね。
いきなり生命情報科学っぽいテーマから入ったんですね。
そうですね。当時は、マイクロアレイDNAの断片をガラススライドに並べたもの。DNAやRNAが相補鎖と結合する性質を利用して、遺伝子がどのくらい発現しているかを計測する技術。のデータを利用してネットワーク推定をやっていました。
遺伝子はお互いに制御したり・制御されたりの関係なので、それぞれを矢印の関係で書くことができます。
たとえば、遺伝子Aをノックダウンしたときに、別の遺伝子Bの発現量が上がったら「遺伝子Aは遺伝子Bの抑制因子」、逆に遺伝子Bの発現量が下がったら「遺伝子Aは遺伝子Bの活性化因子」ということが考えられます。この関係を遺伝子間でつないでいって大きなネットワークにしていくという仕事です。
おお。相関図みたいな感じですな?
 そうですそうです。酵母の遺伝子300くらいを一つずつ欠失させたデータを使ってネットワークを描けないか、ということで取り組みました。
そうですそうです。酵母の遺伝子300くらいを一つずつ欠失させたデータを使ってネットワークを描けないか、ということで取り組みました。
自分でデータ取ったんですか?結構大変な仕事量になりそうな……。
いや、Cellという雑誌に載ってた論文のデータです(笑)。でもこのデータが結構衝撃的で。当時のマイクロアレイはクオリティがそれほど高くなくて、遺伝子の発現量が低いところでは値がすごいバラつくんですけど、この論文ではワイルドタイプを50サンプルくらいやって、統計的にエラーモデルをキチッと作って、遺伝子発現量の差をかなり厳密に議論してたんです。それでこのデータなら信頼できると思って使いました。
遺伝子同士のやり取りって、フィードバックとか迂回路みたいなのがありません?
そうそう。そこがこのテーマのミソです。
たとえば、
AがBを制御している。
BがCを制御している。
とすると、こういう図になります。
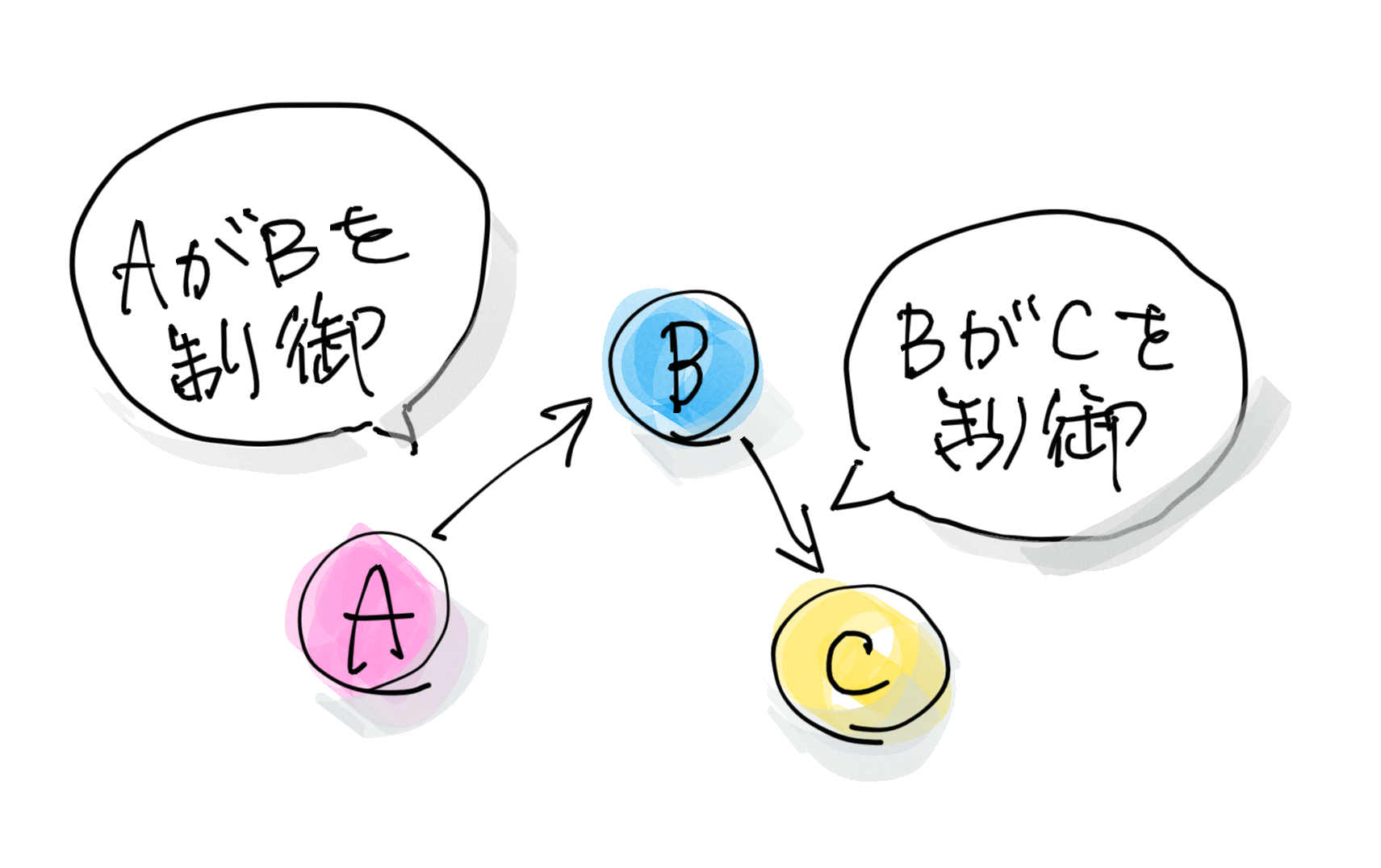
でも、マイクロアレイのデータは、ある遺伝子をノックダウンしたデータなので、BもCも発現量が変動して計測されます。そうすると、実際には存在しないはずのA→Cという矢印が描けてしまいます。でも、本当はないはずなので、これをなんとか消したい。本当にある矢印だけでネットワークを描きたいわけです。
なるほど(ついていけるかな……)
そこでグラフ理論というものを適用しました。僕が利用したのは、ある地点からある地点の間にどういう経路が存在しているか、どのくらいの距離なのかというようなことを調べる方法です。経路探索の一種と思えばいいと思います。
AからCという二つの地点があったときに、この間にどんな経路が存在するかというのを一つずつ確認する作業を行って、ダイレクトな関係のみを抽出するってことをやります。
で、どうやるんですか……?
A、B、Cがあったときに、AがBを制御して、BがCを制御して、AがCを制御してるとしましょう。そうすると、こういう三角なネットワークが描けますよね。AからCにいく時に2本以上の線がある経路を探します。
本数を数えればいいのかな。
まぁ、そうですね。こっちは1本、こっちは2本になります。とすると、A→Cというダイレクトな経路はA→B→Cから生まれたバーチャルなものかもしれない、と言えるわけです。
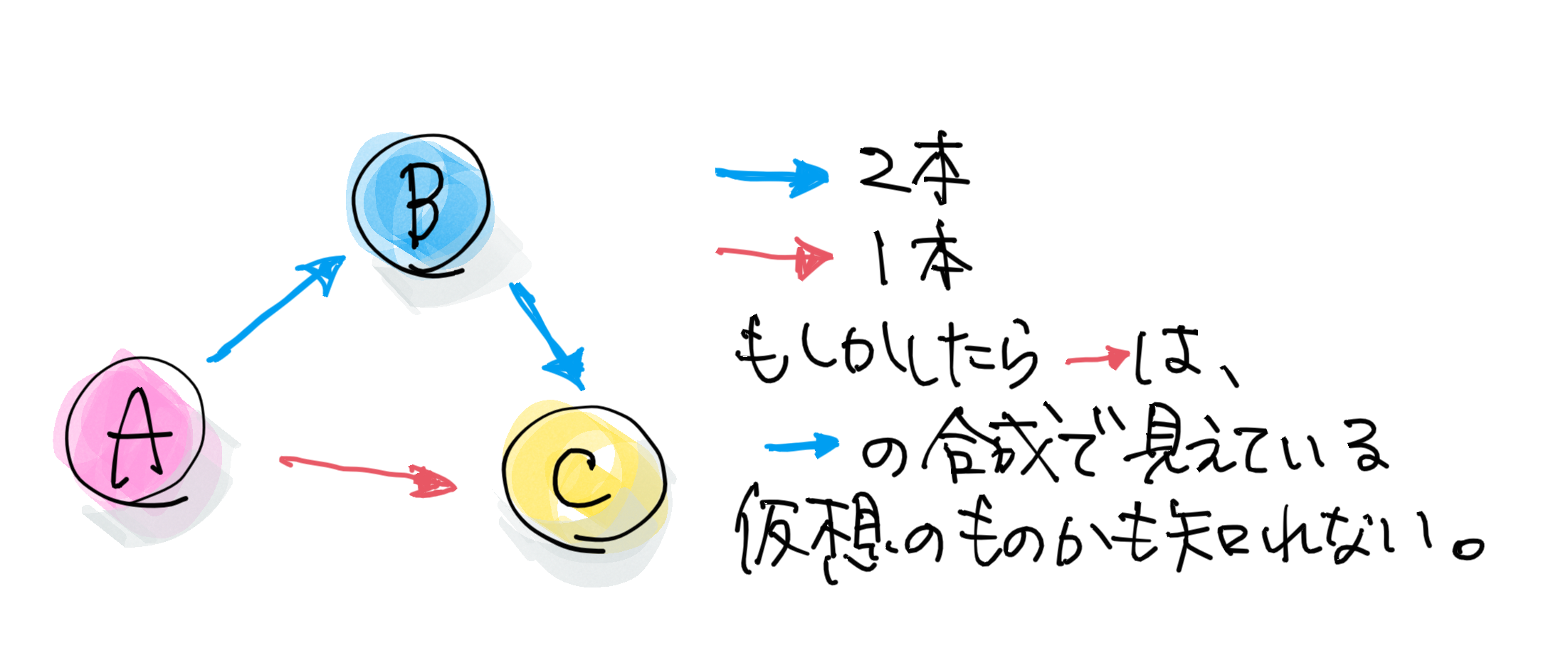
実は、これは活性か・抑制かというのも同時に判定します。活性+活性なら最後も活性になるし、活性+抑制だと期待としては抑制になります。たまに活性+活性なのに最後が抑制になったりする。これはAがCをダイレクトに制御しているはずだ、と考える。
これって総当たり戦?計算量がすごそうな予感。
総当たり戦です。なので、遺伝子数が多くなると計算量はヤバくなりますね。
でも、アルゴリズム的にはキチッとやったので、ちゃんと動きましたけど。
とはいえ、そうやって間接的なものを全部取り除くと、元のデータを説明できなくなるんですよ。
な、なんですと?
これが結構難しい問題で、たとえば、いいスカ?
A→B→CとA→C、C←Bという矢印が書けたとすると、BとCの関係だけに収束するので、A→BとA→Cがいらない、ということになります。そうするとAがぽつんと島になってしまう。Aはどこかに関与しているはずなので、これでは間違っている、ということになります。
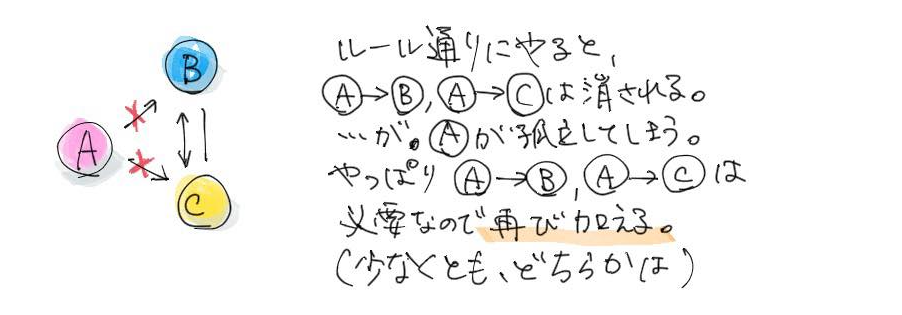
なので、こういうときに、最少の経路を足して、もとのネットワークを説明できるようなものを構築するっていうのが、次にこのメソッドでやることで、たとえばどちらかに1本足せば、Aを組み込むことができるようになります。
ってことは、最初にわーっと全部消して、あとで足すんですね。
そこの足す作業というのが結構エグくて、1本たすのか2本足すのか3本足すのか、みていくんですが、最初は1本ずつ足すってのをやってたんですよ。そうすると、ある数になった時の答えを出すのに計算量が増えすぎて、1年かかるみたいなことがわかって焦りました。
その間に、実は局所的にここだけ考えればいい、という考え方ができることが確認できたので方向転換してアルゴリズムを組み直しました。
このあと、フェロモン応答のパスウェイとかよく知られているパスウェイを使って期待通りにネットワークが書かれているか検証したんですけど、その当時は今ほどデータベースの整備が進んでいなかったのでペーパー論文のこと。paper。研究者間ではペーパーと呼ぶことが多い。を数百本読んで、実際にそのパスウェイがあるか、それを再現できているか、というのを確認しました。
地道な作業すぎる……。いま同じようなことをやろうとおもったらNGSNext Generation Sequencerの略。次世代シーケンサー。旧来のサンガー型と呼ばれる配列解析(シーケンサー)とは原理が異なり、高速に配列解析を行うことができる。データが膨大。のデータになるだろうね。
いやー、エグいっすよ、きっと。いま全然違う仕事してるんで実際のところはわかりませんけどね。
ライフサイエンスの潮流
あ、そうでした。今は、線虫で画像な感じですよね。
当時ね、若造なりに今の世の中ちょっとおかしいなと思ってたんですよ。
研究の流れとして、ゲノムがあって、その次はマイクロアレイがでてきてトランスクリプトームになって、次はプロテイン-プロテインインタラクションタンパク・タンパク相互作用。タンパク質は複数集まって複合体を作って機能することが多いので、何と何が相互作用するのか研究する分野。だ、メタボロームだ、って進んできましたよね。
どんどんどんどん移り変わっていくというか、僕の中では、一つずつがちゃんと終わってないようなイメージなんですよね……。
たしかにね。
今はそれらを統合して新たな知識を抽出しようという動きがあるので良い流れだとは感じています。それともうひとつ。生物の実験って、細胞で実験して、すりつぶして、量このくらいという議論をすることが多いわけですけど、ぶっちゃけいうと「ほんとかよ。」というのを疑問に思ったことがあって。
なんで?
データを扱う側からいうと、あやふやな部分が多すぎるんですよね。
細胞をすりつぶしてDNAをとったりするとき、どのくらい内容物を維持できてるのかもわからないし、そこからマイクロアレイだったらハイブリダイズDNA、RNAは二本鎖を作る性質がある。マイクロアレイの場合は、ガラス状に貼り付けられたDNAと、サンプルから抽出したRNA・DNAとの間で二本鎖を作らせることをハイブリダイズ(Hybridize)という。するわけですけど、どのくらいきちっとハイブリダイズしているのか、とか。そういういろんなファクターでデータって変わってくるじゃないですか。
見ているものがほんとにそうなのか?そんな疑問ですね。もちろん技術が解決してくれることはあるんで、一概にだめというつもりはないんですけど、マイクロアレイのデータをつかっていたときは、そこに気持ち悪さがありました。
もう少し自分が信じられる、具体的には、目で確認しうる現象を捉えたデータを解析したいという気持ちがと言えばいいかな。
情報よりや化学よりで生物を扱っている人たちに共通している気持ち悪さかもしれないなぁ。ときどきそういう話を聞きますね。
表現型を網羅的に
で、線虫の初期胚発生を画像解析で解析・解明するっていうのが今やってることです。
いきなり個体に行ったか。
今だったら、ゲノムとかトランスクリプトームとかには次世代シーケンサーがあって、結構網羅的に取ることができるし、プロテオームとかメタボロームとかは質量分析装置があるおかげで、ある程度網羅的にデータが取れるようになってきてます。ただ、フェノーム表現型を意味するphenoにomeを足した造語。genome、proteomeなどと同じ。表現型を網羅的に解析すること。は、人の目でやってたので、最終的なアウトプットがなかなか網羅的にないというの状態だったんですけどね。
バイオイメージインフォマティクスという新しい分野が確立しはじめていて、生命画像を何らかの処理をして知識を抽出するという動きになっています。
要は見るんじゃなくて、画像を処理することで、何らかの知識を抽出するということです。そうすると、計算機に画像を放り込んだら知識を抽出してくれるので、ハイスループットの方向にいける、という状況になってきています。
カメラの性能も上がってるしね。
ハードディスクの容量も大きい要因ですね。画像データって大きいですから。
で、こういうデータが取られるようになってきたんです。
おおー。これすげー。
こういうのはGFPgreen fluorescent proteinの略。緑色の蛍光を持つタンパク質。1960年代に下村らがオワンクラゲから発見。2008年ノーベル化学賞。他のタンパク質と繋げて発現させることができるため、目的のタンパク質がどこにあるのかをみるときに使われる。とか蛍光標識を使うので蛍光顕微鏡になるんですけど、僕がやってるのは線虫なので、微分干渉顕微鏡通常の顕微鏡では見えない透明なサンプル(細胞など)の構造を、プリズムと偏光板を用いて濃淡をつけることで可視化する装置。灰色に濃淡のついた画像になる。でやっています。
線虫は意外となんでもできる、モデルとしては非常に便利な生物なんですよ。
で、線虫600くらい胚発生に関わる遺伝子が知られていて、RNAiしたときに卵から孵化できるかできないかをみてやると、孵化できなかったら胚発生に何らかの機能を果たしているだろうということが考えられる。
致死性の遺伝子ってことですね。
そうですね。その中でかならず孵化できないはずの351の遺伝子をターゲットにしていて、それだったら、なんらかの異常が出るだろうという目論見です。それを一つ一つRNAiやってデータをとりました。
統計解析をやるためには複数の個体で比較したいので、1遺伝子あたり5セット取る、ということをやってます。
線虫って透明なんで、ステージ上げ下げすると3次元的な画像が取れる。これを一定間隔で最初にとって、そこから画像処理のアルゴリズムを使って。
微分干渉顕微鏡の画像を使って、たとえば核の部分を抜き出していると。
画像情報を数値データに
一旦データが取れればxyztの座標の情報なので、これは計算機上でいかようにも料理できるわけです。
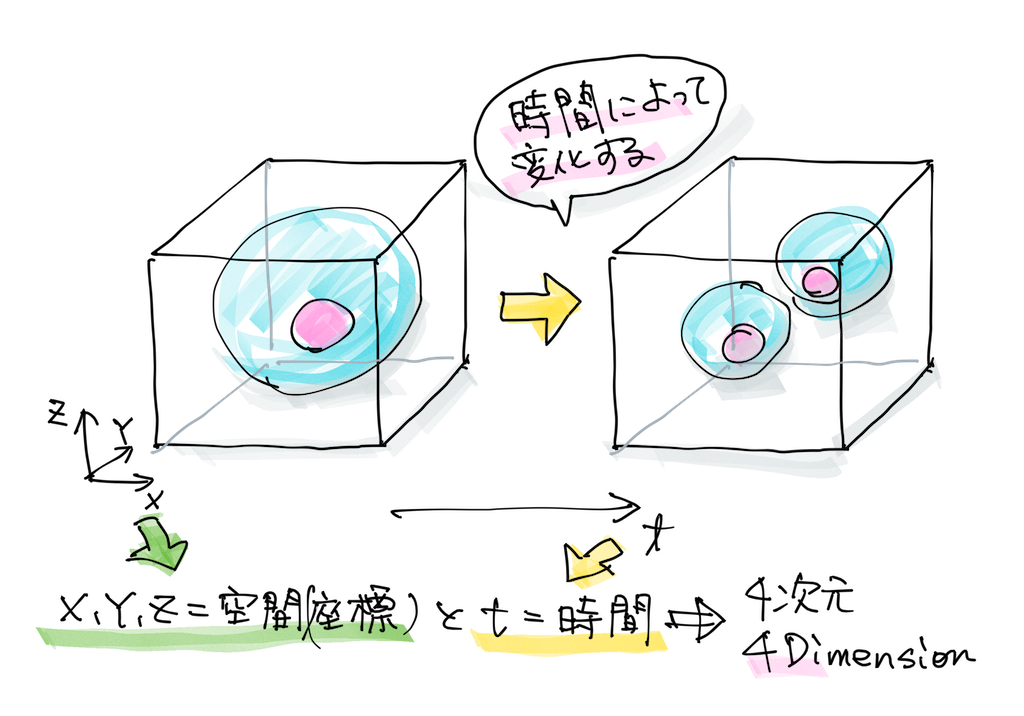
核の体積とか中心座標とか、この核とこの核の距離だったり、それまではものさしで図るくらいしかなかったんですけど、全部データとして出すことができます。ってことは、ぜーんぶ自動で計算ができるようになるってのがミソですね(笑)。
なので、こういうデータを一旦取ってしまえば、あとはいろんな解析に応用できます。なので、いまペーパーにまとめてるんですけど、パブリッシュpublish=出版。論文が学術雑誌や出版社のサイトに掲載されること。されたら画像も公開して、誰かに活用してもらいたいと思っています。
かつてのマイクロアレイのデータのように、ですね。で、どんなデータなんですか?
で、まず最初に2,000くらい、4次元(xyz+t)の微分干渉の画像を撮って、その中には核の情報だったり、紡錘体細胞分裂の際に、別れた二つの細胞へ引っ張る糸状の構造物。の情報だったり、細胞の情報だったり、細胞質の情報だったり、動いているものは方向・速度など、そういう情報も入っていたり、配置も入っていたりします。
さっきお見せしたような蛍光顕微鏡の画像って、たとえば核を染色したりしちゃうと核の情報しか取れないわけですよね。細胞の形とかはわからないわけです。で、核と細胞を取ろうとすると2色必要になるんですよね。なので、せいぜい3色とか頑張ってとるとか、でも3つの情報しか取れないんです。
でも、微分干渉計っていろんな情報が含まれてるんで、この画像さえとっておけばいい。いま、僕らは核と紡錘体の情報しか抽出してないですけど、たとえば、このデータを使って細胞の形をとるとか、そういう人が出てきてもいいと思っているんです。
で、今度は自分でデータも取っているわけだ。
結構エグいっすよ京田節で、「大変」ということを「エグい」という。。
1個の遺伝子をノックダウンするのに3日1セットなんですね。インジェクションを初日、撮影を翌日、3日目に生死チェック。1週間に3遺伝子しか進まないんです。1年50週しかないんで、3遺伝子で50週だと一年に150遺伝子。3年はかかる計算ですね。実際のところは、月曜に休みがあったりすると1サイクルできるかみたいな感じになっちゃいます。途中で装置がトラブルこともありますし。
これまた地道な時間のかかる作業ですね。
画像をとるのに結構時間がかかって、画像処理は計算機にやらせればいいんですけど、1個体分ずつ5台のマシンを並列にして突っ込みます。出てきたものを、全自動でできればいいんですけどエラーもまぁまぁ出てくるんで、今は手でキチッと修正して、使えるデータにしている状態になってますけど、そこもまぁ結構時間がかかっています。
画像から発生のメカニズムを、というのはどういうイメージなんですか?
まずやったのは、遺伝子がどこでどんな機能を果たしているのかというのを見ています。
一個一個RNAiしてるんで、どれを潰しているかはわかってます。
それに対して、野生胚と比べて、いつどこで異常が起きているかというのを見つける。それは計算機で統計的に見つけてくれる。
たとえば、どんな異常なですか?
細胞の核の間の距離って、こんな式で書けます。xyzの座標なんで、計算で全部出せます。
$$d=\sqrt{(x_2^2-x_1^2)+(y_2^2-y_1^2)+(z_2^2-z_1^2)}$$
とすると、3つの核のなす角度とかも計算で出すことができるんですよね。そういった指標をたくさん作って、野生胚とRNAi胚の数値を統計的に有意に差があるかないかっていうのを見てあげれば、たとえば、この表現型、この角度に異常があるかないかというのを全てのRNAi胚で確認することが自動で統計的に検定することができる。
個体としてはどのくらい異常がでる?あれ?どのくらいの時間データを取ってるんでしたっけ?
初期発生のステージで、受精してから1時間くらい、8細胞期までです。20秒間隔で、xzyの3次元情報を画像としてとります。それと孵化したか、していないかの情報ですね。
面白いのは、胚性致死の遺伝子を狙ってRNAiをしているんですが、たまにちゃんと生まれてくるものもいるんです。
データ・ドリブンのライフサイエンス
だめそうなやつも軌道修正しちゃうと。
いまはまだそこまでできていないですけど、変動を元に戻すメカニズムがどういうものがあるか、というところまで発展できたら、と思っています。
どうやって画像の情報から生物学的なことを語っていくんですか?
たとえばセルサイクルで説明しましょうか。線虫の場合は、間期だと球に近くて、分裂期になると潰れた形になるので、球形度という指標で細胞の状態を決めることができます。で、時間の情報も持っているので、どの細胞がどのくらい間期で、どのくらい分裂期か、というのも数値で出すことができます。同時にRNAiのターゲット遺伝子はわかっているので、どの遺伝子をノックダウンした時に、どの細胞の何細胞期に、間期が異常に長い、というようなことが言えるようになります。
これまでも感覚的に、こういうときは間期が長そうだ、みたいな議論はありましたけど、それを定量的に議論できるようになった、というとこですね。
なるほどなぁ。その「特徴」ってどのくらいあるんですか?
400くらいですね。で、特徴を横、遺伝子を縦にとってやると、こんな図になります。色が塗られているところがなんらかの異常があったところです。
で、これ、どうするの?
まずは、過去に報告されているものとの比較を行いました。2,000以上新しい特徴を見つけています。3次元空間の把握は人間には難しいので、そういうのも結構ありますね。特に顕微鏡を使って見ていると、深さ方向(z)ってステージの上げ下げでしか見えてこないので、認識しづらいし、比較はもっと難しい。分裂方向がz軸方向だと、認識は難しいでしょうね。そういうのが数値として出てくるので、客観的に異常を検出することが可能になります。
今は8細胞期までですけど、もっと長く見たりすることはやらないんですか?
細胞がどんどん小さくなっていくので、微分干渉系では難しいだろうとは思います。ジョン・サルストンというノーベル賞をとった研究者が1,000細胞になるくらいまで追跡しているんですけど、よく追跡できたなと思います。1980年くらいですね。VHS若い読者は知らないかもしれないので、一応解説。カセットテープ型の動画記録装置。VHSとβの2種類があったが、VHSが主流だった。テープなので、繰り返し再生するとテープがへたってくる。1000細胞を追跡するには、きっと擦り切れるまで見たに違いない。を使ってやったみたいです。なかなか強烈です。
間違ってる部分もあるかもしれませんね。にしても、研究者ってすごいな……。
「一部逆転することがある」というような但し書きが付いている部分もあります。
でも、この追跡の過程でアポトーシスが見つかって、それでノーベル賞をとったんです。
たしかRNAiも線虫でしたが、アポトーシスも線虫で見つかったんですね。
そんなのリネージ細胞系譜。細胞が分裂し、分化していく系譜のこと。全部追っかけてないと見つからないと思います。偉大な発見ですね。
新たに表現型を検出したいってときに、ビデオ全部見直すなんてことはしなくて、一旦定量化していると、特徴抽出するプログラムだけで高速に再スクリーニングすることができるってのも僕らのやり方のいいところです。
画像から分子メカニズムへ
で、この表、どうするんですか?
クラスタリング似た特徴を持つものをグルーピング化していく手法。ってのもやってます。いろんな表現型の特徴をとってるんけど、それをクラスタリングしてやると24くらいのクラスターに分けることができます。言い換えると、8細胞期までに起こり得る表現型のパターンは24通りということが言えます。それぞれがなんらかの細胞プロセスに対応しているんじゃないかと思っています。
ちょっとイメージわかないんですけど……。
たとえば、どっちの方向に分裂するか、タイミングとかですね。
関連する遺伝子とかもわかる?
そうです。RNAiしてるので。このリストの中の一つ一つ遺伝子を調べていくと、どういう細胞プロセスに対応しているかというのが統計的に出てきます。とすると例えば2つの遺伝子があって、どちらもある同じプロセスに関係しているといわれているけれども、僕らの解析では違うクラスターにある。とするとつまり、実は少し違うプロセスに関与している可能性があるわけです。
24クラスターで、ちゃんと見てやれば発生プロセスが理解できるんじゃないかと。
さらにここから、細胞のプロセスがどういうメカニズムによって達成されているかというのを見ようと思って、相関解析をやってます。表現型と表現型の相関なんですけど、全て数値化されているので、これも数字として扱うことができるんです。そうすると、ある特徴とある特徴の間に相関があることがある。これは非常に重要です。特徴同士が因果の関係になっているか、あるいは別にエフェクターがあるということを示唆しています。共通するメカニズムがあるだろう、ということです。88,000通りの組み合わせのうち3,000くらいに強い相関があることが見えてきました。そんなに多くはないですけど。
ちょっとだけ具体的にお願いします……。
ある細胞と隣の核の軸の角度が、細胞分裂の方向を決めているのではないか、とかが出てきます。でよくよく調べてみると、隣の細胞からシグナルが出ていて回転する、という報告があって、それを予備情報なく抽出できた、ということです。
全部は見てませんが、3割程度はどの生物学的プロセスに対応しているかというのが未知のものになりそうです。
つまり、この3割には新しい発見のタネが眠っている可能性が高い。しかもデータを公開すればこれを誰でもできるようになる、というのが大事なところです。
なるほどー。AIも含めて画像解析とかの話は、結構従来の話をトレースしただけじゃないか、と素人的には思ってしまうこともあるんですけど、やっぱその先があるんですね。
たしかに、これまで生物学者がやってきたことをどのくらい高速でやるか、くらいの話に思われがちなんですけど、定量データが得られているからこそ発見できる、あたらしい知見というのがあるんです。
さらには、相関がある場合に線を引いていくと、割とでかいシングルネットワークになっていくんですけど、特徴を継承していって発生が進んでいくというのを初めてきちんと表現できた図になるかな、と。
ところで、いままでの話って、さっきの3年かけてとったデータを解析してるだけってこと?
そうです。それでこのくらいの知見がどんどん出てくるんです。そこがいいとこです。
もしかしたら宝の山かもしれない、ってことですね。
そうですね。まだまだいろんな解析ができると思います。
画像をいじる必要もない。
いらないですね。数値データがあるので。
さっきの回転の話とか詰めていくんですか?
それはそこに興味のある研究者がやるのがいいと思っています。やっぱり全部はできないですし。やはりデータはオープンにしていくのが良いと思います。
サイエンスの中心が移動していく
データ駆動型データが大量に取得できるようになっているので、(すこし乱暴な言い方をすれば)とにかくデータをとってそこから考えるという考え方。データドリブン。のサイエンスって、とりあえず解析してみて初めてわかることも多い。僕は何かの新しいメソッドを開発・提供していくというのが好きですね。博士の時も、その当時はベイジアンネットワークとかブーリアンネットワークを基盤とした手法が主流でそれを適用しましたというのが多かったったんですけど、僕は違うアプローチで、遺伝学の考え方プラスグラフ理論だったり、新しいことを作ったりしてたわけです。
いまも表現型というだけでは当たり前にできることになってきています。それでも結構苦労しましたけどね。それよりももっとアドバンスな開発をやるというのが大事にしているスタンスでいます。
まぁ、面白いことができればいいですね。あと必ずしもアカデミアじゃなくてもいいかもしれないとも思ったりしています。
アカデミアは面白いんですけど、僕がやってるような画像解析とかは、これからもアカデミックがNo1でいる保証は多分ないと思っているんですよ。GoogleとかがAI使って、病理診断とかやり始めてますよね。そうすると、あっという間に民間の方がトップレベルに行ってしまうことも考えられます。そうするとアカデミックでいる必要もないかもしれないとずっと思ってます。
たしかに、RNAiや変異解析など数を稼ぐ必要があるものは、カネとヒトを投入できるところが強くなりますよね。
そうです。顕微鏡をわーとたくさん並べたら、僕らが3年かかってとったデータくらい、あっという間に取れちゃうかもしれない。いま台数が限られているのでシリアルにやるしかない。それをパラレルにやられてしまうと、あっという間に駆逐されてしまう。
物量勝負になっていきそうですよね。データを持っているところが、新しい解析だったり発見だったりをする可能性がありますよね。

そうするとデータをもってるところが勝ちですからね。今のGAFAGoogle、Amazon、Facebook、Apple。IT界のビッグ4。Microsoftを加えてGAFAMということも。これに比肩できるのは、中国のBAT(百度、アリババ、テンセント)くらいしかないのではないかと言われていて、日本は大きく水を開けられている。とかがわかりやすい例ですよね。
じゃぁ、次お会いするときはg〇〇gleの京田さんってことでw
編集後記
「いやー、濃かったー」というのが録音の最後の言葉でした。がっつりお話が聞けて面白かったという自己満足な感じではありますが。データドリブンの研究の方向が強まってきている感がありますが、やはりGAFAが強くなっていくのだとすると、アカデミアの存在意義が問われているのかもしれないな、と改めて思いました。
京田さんのような仕事のやり方ができるのも、データベースがしっかり整備されているからに他ならない、ということも改めて認識しました。
ちなみに、少し古い2014年の動画だけど、どんな映像になっているかわかりやすいのがこちら。
