ミクロとマクロを繋いで新しい生物学を
理研BDRにはいろんな研究をしている人がいるんですが、今回インタビューする久米さんはその中でも「何やってるのかわからないヒト」なんじゃないかなと思っています。なので、何を研究しているのか、ネホリハホリしてきました。(聞き手:薬師寺秀樹)
さまざまな網羅的な情報を取り扱う
久米さんとはいろんなところで会うんですけど、内容が多岐に渡っているので、実際のところの研究の内容が実はよくわからないんですよね。
僕もよくわからないんですよね。何やってるんでしょうね。
領域広そうですよね。どこに行っても久米さんの知り合いに会うって聞きますし。
それは脚色が入ってますよ……(汗
研究テーマとしては?
理研に着任した当初は疲労の研究からスタートしました。今は、さらに広げて健康科学や未病、あと最近では電子顕微鏡などのイメージング技術開発、画像解析、白血病、腎臓病なんかも研究のトピックスになってて、「生物x情報科学」全般を扱うようになってきてます。
健康や疲労は、もともと興味があったんですか?
そうですね。学生のころは違うことをやっていたんですが、生物はいろんなところで絶妙に繋がっているんですよね。
疲労とか健康とか未病とか、漠然とした状態だとおもうんですが、どうやって扱うんですか?
そもそも、生物の状態は、ひとつのデータで記述することが難しいところがあります。たとえば、血糖値だけでその人の全身の状態を記述することは到底できないですよね。
そのため、いろんな角度からのデータを取って、網羅的なデータで生物・生命の状態を表現して理解する、ということが大事だと思います。
網羅的なデータって、結局何を示唆しているのかわからなくなりそうですけど?
そうなんですよ。だから、網羅的なデータのなかに見え隠れする発見の原石をどうやって炙り出していくかとか、得られたデータをどうやって客観的、かつ分かりやすく定量・可視化していくか、そういう解析のストラテジーを考えるところがよりいっそう大事になります。
実際のところ、どうやるんですか?
例えば、よくやるのは、たくさんのデータを同時に扱う多変量解析ですね。あと、今だったら人工知能(AI)の手法で、機械学習・ディープラーニングを使うことも多くなりましたね。
現在の生命科学では、いろいろな種類や深度のデータが取得できるようになってきて、それと最新の情報科学をミックスさせることで、新しい生物学的な発見が生まれるんじゃないか、と考えています。
いきもんを理解するのにはまだ全然足りない
久米さんのいう「新しい生物学」ってどういうイメージですか?
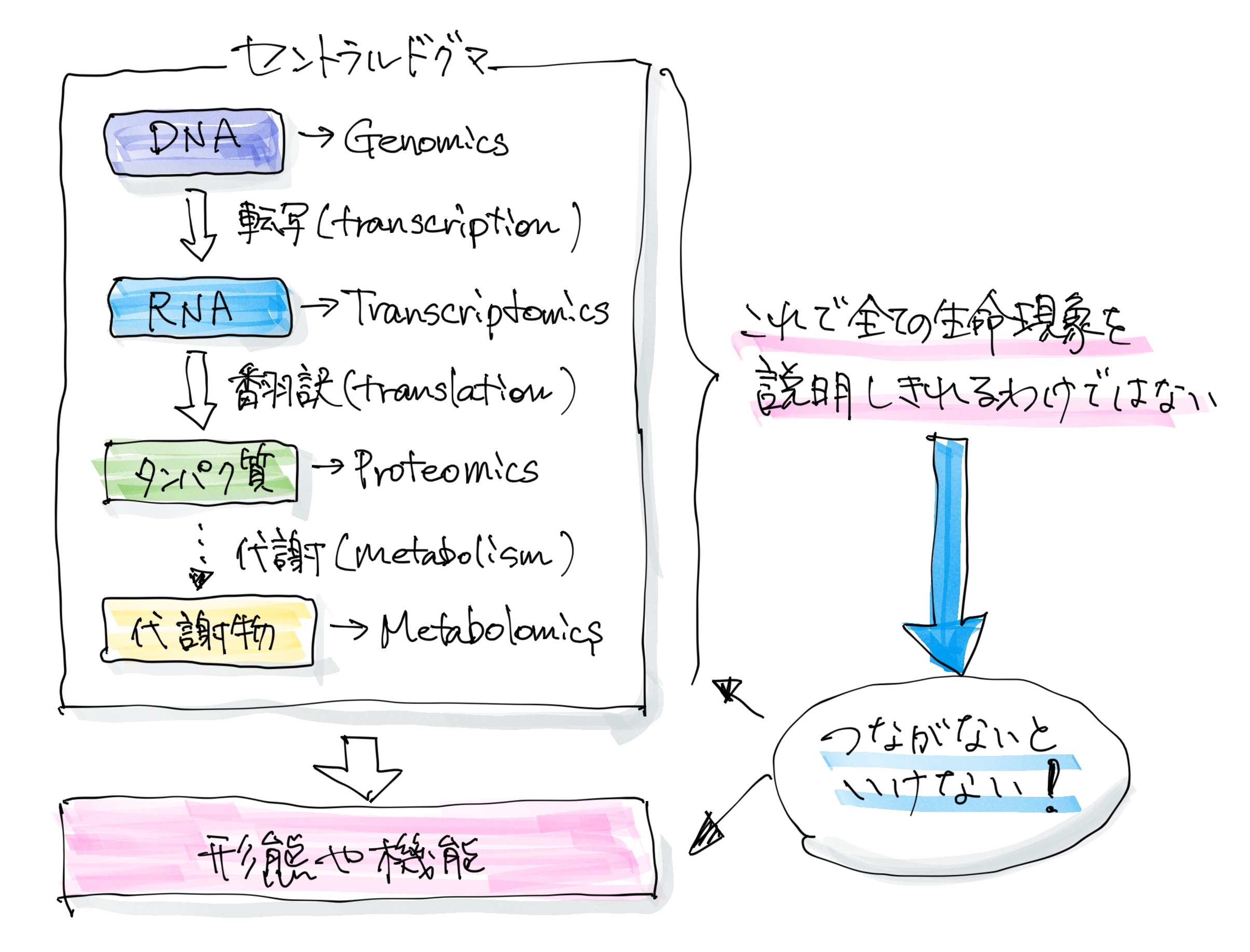
たとえば、生物学には、セントラルドグマDNA→mRNA→タンパクという一連の流れによって、生物が規定されているという考え方。現在は、エピジェネティクス、ncRNAなども含め、新しい概念が登場してきていて、かならずしもセントラルドグマで説明しきれないと考えている研究者もいる。という考え方があります。DNA、RNA、タンパク質の順で情報が伝達される、という概念ですね。最近ではさらに概念が拡張されて、代謝物まで含めることもあります。
我々は、ゲノミクス、トランスクリプトミクス、プロテオミクスといった網羅的解析(オミックス〇〇-omeを取り扱う学問領域を〇〇オミックスという。ゲノムの場合はgenome+omicsでgenomics。RNAの場合は転写物なので、transcript+omics=transcriptomicsなど。)の研究として、生命科学の授業で教わるんですが、あれってもっと別の領域があるんじゃないか、と思うんです。
その一つは形態学と思っています。細胞内の遺伝子発現とか代謝物とかが変化することで、最終的な表現型遺伝的な要因(=遺伝型)に対して、形や機能などとして現れるものをいう。英語だとgenotypeとphenotype。遺伝子検査のことをジェノタイピングというのはこの遺伝型を調べるから。として、形態の変化として現れることがよくあります。
モルフォロジー(morphology)構造などを含む形態。細胞や組織などカタチを持っているものについて研究する分野。というやつですか?
そうです。モルフォロジーですね。
網羅的にみるって、どういうイメージですか?
多くの場合、研究者はある仮説を持っているわけで、研究者はその仮説の検証に関係してそうなところしか実験しません。簡単に言ってしまうと、自分の見たいところしか見ないんですよ。
遺伝子発現解析を例に挙げると、以前はPCRPolymerase Chain Reaction=ポリメラーゼ連鎖反応。DNAが相補的な配列を複製する性質を利用して、目的の配列のDNAのコピーをたくさん作る技術。測れないくらい微量のDNAの量を増やすことで、検出できるようになる。これにより分子遺伝学が劇的に進んだ。1993年ノーベル化学賞。で自分の見たい遺伝子だけ増やして、その遺伝子がどうなるかを調べる、みたいな研究が普通でした。ところが、ここ10年くらいで次世代シーケンサー2000年すぎころから急速になってんしたDNA配列装置。この技術の登場で、遺伝子の発現解析が劇的に網羅的になり、情報量も多くなった。現在は、1人の全ゲノムが数日、1,000ドルで解析できるようになってきている。といった新しい技術が普通に使えるようになってきました。
その結果、網羅的なデータを取って恣意的な部分を排して、新しいことを探れるようになってきた。DNA、タンパク質、代謝物などのオミックス研究でも、実際そういうアプローチが主流になってきています。
形態学や画像解析は、言ってみれば、まだPCRの時代なんです。自分が見たい視野の範囲でしか解釈を構築することができません。そこで、細胞をもっと高分解能で見たり、組織全部とか体全部を見たり、もっと網羅的にイメージングして解析できるアプローチがあれば、また違った発見が起こるのではないか、と考えています。
難しそうですね。
難しいですよ(笑)。
ただ、幸運なのは、イメージング手法やコンピュータのハードウェアもだいぶ技術が進んできています。例えば、電子顕微鏡の撮像では一度に100ギガバイト以上の画像データが取得できるようになってきましたし、テラバイト以上のデータをラボレベルのパソコンでも扱えるようになってきています。私が学部生のときに買ったパソコンのハードディスク容量は40ギガバイトヤクシジが学生の頃は100メガバイトで十分大きかったので、10年で大きく進展したことを改めて痛感……。でしたから、それを考えると、すごい進歩ですよね。
なので、これまでは自分の好きなところだけ撮っていたのを、客観的、かつ、網羅的に撮ってやると、恣意的なところが除かれて、もっと生物の理解が進むんじゃないか、と思っています。
ところで、どうなったら生物を「理解した」と言えると思います?
どうでしょうね……。ひとつは、インプットからアウトプットまでの因果関係のパスウェイがすべて把握できたら理解したと言えるんじゃないですかね。
たとえば、病気になると言ったって、その因果関係はわかっていないこともあります。その間がすべて繋がったら、理解したことになるんじゃないですかね。
いま得られている情報だけで十分なの?
いま得られている情報だけで生物を理解するのが難しいのは、やっぱ、何かが抜けてるってことかもしれません。今ある情報だけで十分なら、全ての病気は治せるわけですから。病気が治らないってことは、何かの情報が抜け落ちているんでしょうね。
科学者として、そこを埋める努力をしないといけない。そうしないと病気の理解とか、生命の理解なんてところにはたどり着けないわけですから。
何が足りないんですかね。
正直なところ、正しい答えはわからないですし、何が足りないか分かれば苦労しませんよね(苦笑。ただ、情報の一つとしては、さっきお話ししたモルフォロジーもあるんじゃないかと思います。
いまのところ、組織の形態とか表現型とかの「マクロの情報」と、DNAやRNAなどの「ミクロの分子情報」が、どこまでリンクしているかというのもよく分かっていません。まずは、これをつなげるところが大事だと思っています。
あとは、時間ですね。生命現象は時間経過で変わっていくので、4D(4次元)生物は3次元の存在であり時間によって変化していく。これをx、y、zとt(時間)の4次元で理解しようという流れになっている。顕微鏡の画像の多くは3次元の立体データとして取得されていることも多く、この3次元の情報の時間経過を追うことで、4次元情報を取り扱うことになる。の情報として、どうやって繋げていくか、ということも重要だと思っています。
生化学者のような、情報科学者のような
ところで、理研にくる前はどんな研究をしてたんですか?
大学の時は、タンパク質・酵素を使った生化学の研究をしてました。タンパク質の精製とか、酵素化学実験とか、低分子化合物との結合実験とかですね。研究テーマとしては、プロスタグランジンD合成酵素(L-PGDS)というタンパク質の機能解析とか、生物学的な意義についての研究をしていました。
普通に聞いてる感じだと、生化学の人ですよね。それがなぜ情報科学っぽい方向に?
生化学者ですって(笑)
キッカケはその次の研究ですね。当時所属していたラボは、このL-PGDSを薬の輸送システム(DDS : ドラッグデリバリーシステム)に使う、という研究をしていたんですよ。
ほー。どんなデリバリーなんですか?
薬の候補分子の多くは、疎水性が高くて、水に対する溶解度が低いんです。
細胞の膜成分は主に脂質でできているので、疎水性が高い化合物は細胞膜を透過することができるんですが、水に溶けない。水に溶けないと分子同士が凝集してしまって、そもそも届けたい細胞のところまで届かない。逆に、水溶性が高い化合物は水に溶けるけど、細胞膜を透過できなくなる。薬は、水に溶けて、油にも馴染むという、相反する性質が必要なんです。

っとすると、生体の分子とはどう違うんですか?
グルコース(単糖)とかの細胞にとって大事な物質は、細胞膜に特定のトランスポーターがあるので、すごく水に溶けやすくても、トランスポーターを介して細胞膜を通過するんです。
でも、低分子薬の多くは人工的に作り出した化学物質とか天然の化学物質なので、細胞はそれに応じたトランスポーターを持っていないわけです。なので、基本的には、薬の化学的な性質で細胞膜を通るか通らないかが決まる場合が多いです。
L-PGDSタンパク質を使うとどんないいことがあるんですか?
溶解度が低い薬は、そのままだと凝集して、目的の細胞に届かないので、溶解性を持たせる必要があります。そこで、大腸菌などで人工的に作ったL-PGDSで薬剤を抱合してやるというアイデアです。L-PGDSは樽(バレル)型の立体構造をしていて、タンパク質表面は親水性で水に溶けやすい性質なのですが、樽内部は疎水性で、溶解度が低い化合物を結合する性質があります。そのため、本来、凝集するような薬もL-PGDSで抱合させることで、溶解性を高めることができます。
なるほどねぇ
その次に、L-PGDSがどの形状の薬との結合に適しているか、という研究に取り組みました。現在、数千種類くらいの承認薬があるんですけど、それらの化学構造をデータベースから取得して、in silico(コンピュータ上で仮想的に)で、L-PGDSとドッキングシミュレーションさせました。
ようやく情報科学っぽいところに入ってきた。
ミクロとマクロを繋ぐ
先ほどの「間をつなぐ」というのは、インプットとアウトプットの間を埋める作業だと思うんですが。
薬を飲んだら病気が治るとか、そういうインプットとアウトプットはわかりやすいですね。でも、その間は抜けていますよね。まだまだ、その間をミクロからマクロまで説明することは難しいところです。
いまは、実験動物だけでなくて、ヒトを研究対象とするのも良いかなと考えています。
というのも、ヒトだと、病気の情報、健康診断の情報、睡眠・生活習慣などを含めた様々な情報が手に入るわけです。最近だと、ウェアラブルのセンシングデータとかも使えますしね。あと、腎臓領域も面白いと考えてて、他の研究者とチームを組んでいます。
どうやってつなげるイメージですか?
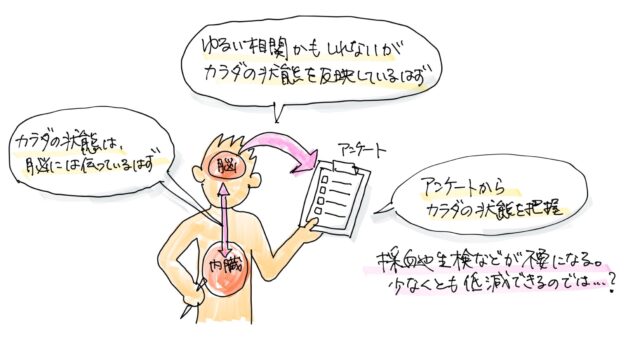
健康計測のデータと、分子情報とか形態画像のデータを繋げようとしています。最近は、健康計測とその人の血球の電子顕微鏡画像をつなげるという研究にもチャレンジしています。
血球の画像データとその人の健康データを繋ぐってこと?
繋がったらおもしろくないですか?
そりゃそうですけど、なんかイメージわかないなぁ。
アンケートで体の状態がすべてわかる!?
いままで人間が考えても到底繋がらなかったことが、人工知能(AI)だと、その相関性を見つけて繋ぐことも実際にできつつあります。
もっと踏み込むと、主観的なアンケート結果と生物学的な情報がリンクするかもしれない、と思っています。
風が吹けば桶屋が儲かるみたいな話ですね。
自分が健康だと思っている人の方が長生きするという疫学的な調査結果があります。そうすると、もしかしたら自覚的な情報というのは結構バイオロジカルな情報を反映しているんじゃないかと考えられます。
生体の現象は独立事象ではないので、例えば、味の好みとか味覚の変化だったり、異性の好みだったりとか挙げるとキリがないですが、そんな、何気ないアンケートでこの値にチェックしたという情報は何かしら生体情報とリンクしているかもしれません。
「アンケートでこういう回答をする人は、こういう疾患があるかもしれない」なんてことができるようになるかもしれないということですね。
そうですね。もちろん科学的な検証は必要なんですが、最終的にはアンケートですべての体の情報が反映していたら、面白いですよね。というのも、アンケートとかの主観的なデータは、脳から吐き出された情報なので、体の状態をゆるいかもしれないけど反映しているはずだと思うんです。
自覚的に肝臓が悪い気がするんですけど……。
自覚があるんなら大丈夫じゃないですか?そのうち病院行くでしょ?そういう人は長生きするという疫学的な報告がありますよ。
AIはどう使うんですか?
脳はものすごくゆる〜くしかアウトプットできないと思うんですけど、このゆる〜い相関性をAIで解明できるんじゃないかと考えて研究を進めています。
たとえば、今日は夕方に目が霞んだとかも、実は、何かのシグナルかもしれないですしね。あとは、体の中で、がん細胞がある程度増殖していたとしても、脳はがん細胞があることに気がつかない。これには、いろいろな分子メカニズムがあるんでしょうけど。実際、がんの早期検査を推奨するのはそのためです。でも、もしかしたら、脳はがん細胞があるってことを検知して、何かのアウトプットを出してるかもしれないけど、我々の意識レベルでは全然理解できていないのかもしれない。

なるほど。自分の体をセンスして、状態がわかるようになるといいですね。
理研ベンチャーの株式会社Kokoroticsの取締役もやっているんですが、この会社では主観的気分測定ツール「KOKOROスケール」を用いた評価システムを提供しています。気分はあくまで主観データなんですが、お話したように、脳は体の状態をセンシングしているはずで、それが1つのアウトプットとして、「気分」とか「情動」として現れるのではないか、と考えていて、それが実証したいと考えています。
編集後記
本文には掲載をしませんでしたが、う●この色の話があったり、知識の幅が広くて感服。(興味のあるかたは、「ビリベルジン」「ビリルビン」などで検索してみてください。)「新しい生物学」というのは、多くの生物学者が考えているところですが、情報科学というのがキーワードになってますね。
