ドキドキ!スクリーニング!
今回お話を聞いた大字さんは、丹羽さんと同様に森田さんからご紹介をいただきました。所属している平谷研はエピジェネティクスの研究をしているというイメージだったので、遺伝子発現のコントロールに関わるDNAの化学修飾のお話しかと思いきや、まったく違うアプローチだったので、ちょっと意表をつかれました。そして、いきなり名前を間違えてスタートしてしまいます……。(聞き手:薬師寺秀樹)
核内のDNAの構造
「だいう」さん。今日はよろしくお願いします。
「おおじ」です……。
げ?じゃ「宇(う)」じゃなくて「字(じ)」? すみません……。
慣れてますので、大丈夫ですよ。
(気を取り直して……)早速ですけど、どんな研究をしているんですか?所属ラボから察するとエピジェネティクスを一細胞で顕微鏡で観察とか、そんな感じですか?
私は顕微鏡というより、FACSFluorescence-activated cell sorting。
蛍光標識した細胞を一つ一つ分けて回収する装置。をよく使います。核内のゲノムDNAの構造に注目しているので、普通の顕微鏡では見えないんですよ。電子顕微鏡を使えばクロマチンの構造は見えるんですが、それでもピンポイントに見たいゲノム領域を探して観察することはすることはできないので。
エピジェネティクス遺伝学=ジェネティクスに対して、「上」という意味の「epi」がついてエピジェネティクス。遺伝子の発現などのコントロールをしているのが何かを探る学問。で核をFACSで見る、と言われてもちょっと繋がらないです……。
そうですよね。DNAのメチル化とかヒストンのメチル化DNAやDNAが巻き付いているヒストンというタンパク質に、メチル基などの化学修飾がされたり、されなかったりで、遺伝子のON/OFFがコントロールされている。とかではなく、核内の構造を制御するものが何なのかを見つけたいというスタンスで私はやっているんです。DNAの三次元構造ですね。
ほどけているときにこうなったりこうなったり(身振り手振り)する?
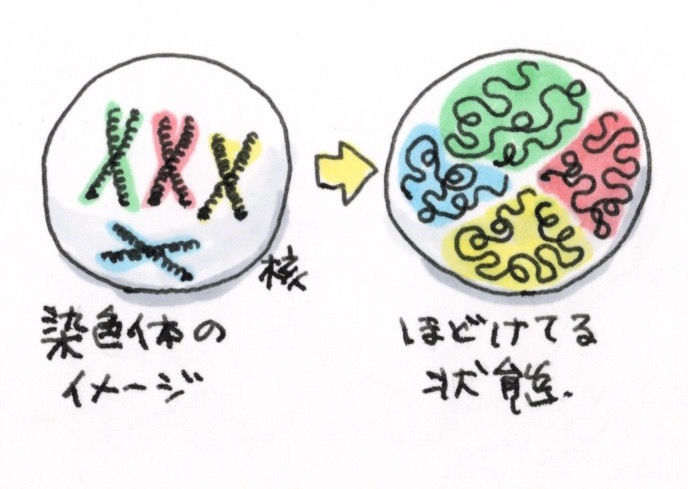
そんなイメージです。
DNAって染色体のイメージが強いですけど、ずーっとあのまんまグルグル巻きになってたら、使いたい時に使えないですもんね。
そうですね。染色体は細胞核の中ではほどけている状態になっていることが多いんですけど、ゲノム上では遠く離れているDNA領域同士が空間的に近づいて一緒に働くことがあるんです。
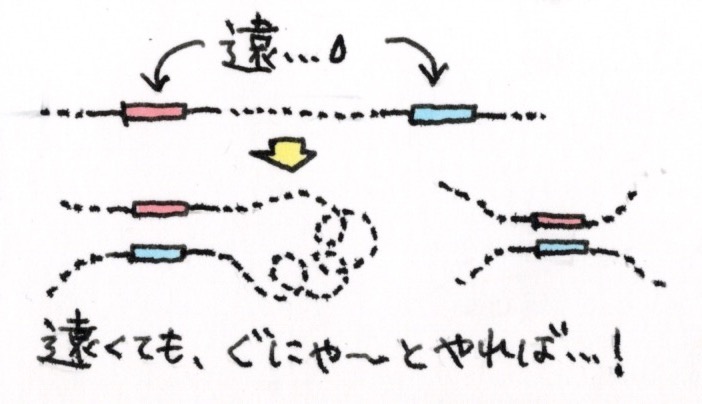
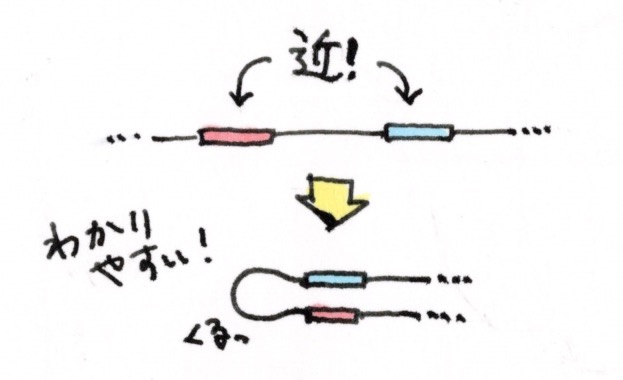
例えば、ある遺伝子と、その発現を促進するエンハンサーが、一次元的なゲノム距離では数百kbと遠く離れているのに、三次元的に見ると、ぐにゃっと折れ曲がっていて隣にいたりします。このゲノム上では遠くにいるけれど、働くときには空間的に近くにいるDNA配列のユニットがTADs(Topologically Associating Domains)核内でDNAが空間的に相互作用している塊。図参照。というボールみたいな塊を作っていて、塊同士で相互作用しながら、核内に収納されているんです。
ほぇー。
で、その塊のうち、活性が高いものをAコンパートメント、低いものをBコンパートメントと呼んでいます。必ずしもAコンパートメントの中にある遺伝子が全て活性化されているか、というとそうでもないんですが、概ねそんな塊です。
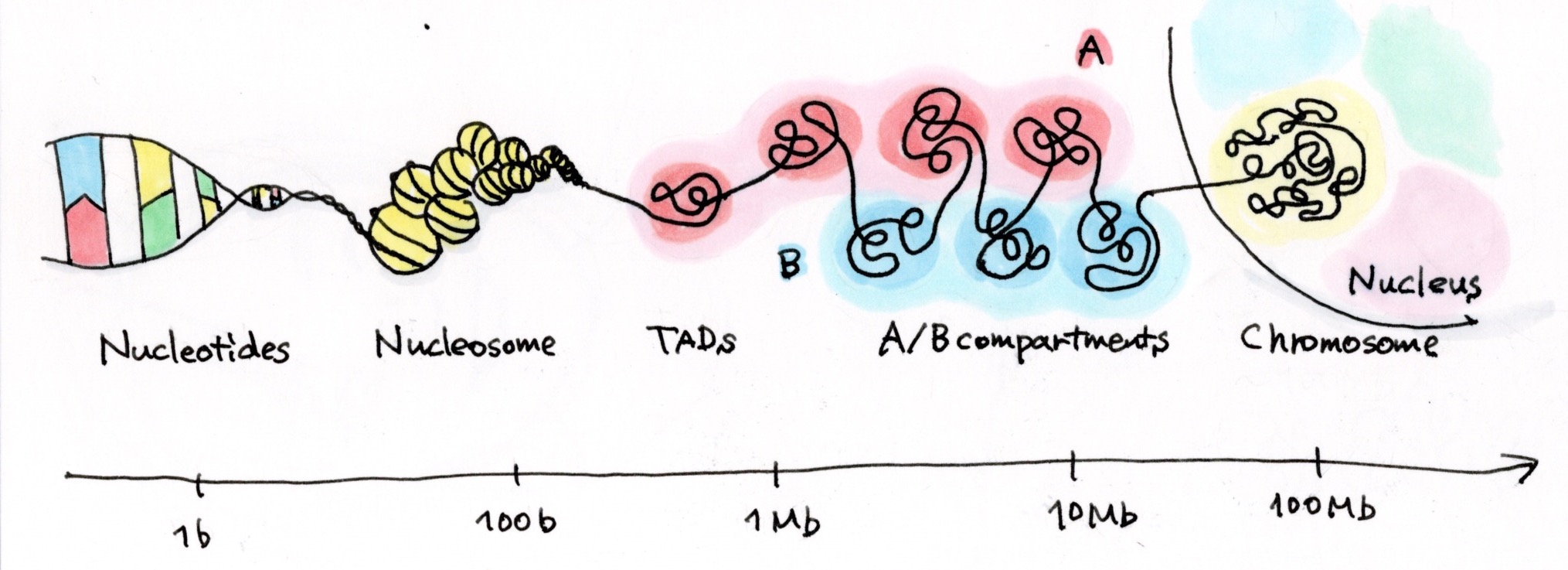
ってことは、同じ個体だとすべての細胞が同じゲノムを持っているけど、例えば肝臓の細胞と心臓の細胞は活性化されている遺伝子が違うはずだから、核内の構造も違うんですか?
そうなんです。この塊の配置は細胞によって変わるんですよ。だから、細胞によって、この細胞ではその遺伝子はBコンパートメントにいるんだけど、違う細胞ではその領域はAコンパートメントにいて、そこでは使われている、というようなことがあると思います。それを制御する何かがあると思うんです。それを探ろうとしています。
複製するときの順番
もともとこういうテーマでやってたんですか?
うちのラボは少し特殊で、核内配置を直接解析するのではなくて、DNAの複製タイミングを切り口にしてDNA三次元構造にアプローチしています。。
DNA複製をするタイミングってゲノム上で決まっていているようで、これも細胞によって変わるんです。DNA複製は細胞周期のS期というタイミングで起こります。ただ、このS期は時間が長いので、早い段階と遅い段階で複製される領域が決まっているんです。
この早い遅いのプロファイルを取ってみると、実はさっきのコンパートメントのプロファイルにすごく似てるんですよ。Aコンパートメントは早い、Bコンパートメントは遅い、と言う感じで、相関係数は0.9くらい。
それは、相関がある、と言っていいですね。
ということで、遺伝子改変をした細胞をたくさん作って、複製タイミングがおかしくなった細胞をスクリーニングで割り出せば、イコール「コンパートメントもおかしくなった細胞」と言えるはずなので、コンパートメントを制御している遺伝子というのがわかるんじゃないかというのがプロジェクトの全体なんです。
ちなみに、それとmRNADNAからコピーされるRNA。このmRNAの配列を元に、タンパク質が合成される。の発現とかが相関している、みたいな感じなんですか?
そういうこともあるかもしれません。先程言ったとおり、Aコンパートメントはクロマチン活性が高い領域なので、その中に含まれる遺伝子発現の活性に関わっている事も考えられます。とは言っても、100万塩基対とかいうスケールのコンパートメントと、せいぜい数十〜数百塩基対の遺伝子では制御のレベルが違うと思うので、単純にAコンパートメントだからmRNAが発現する、という相関の仕方ではありません。
で、FACSはどこで出てくるんですか?
まだもうちょっと説明が要りそうです……。
TADsやコンパートメントを解析するのには、Hi-CというDNAが構造的に近い部分を抽出して解析する技術があるんです。でも、わたしのプロジェクトでは状況的にHi-Cを使うのが難しいんです。
スクリーニングでHi-Cを使うとなると、何か一つ遺伝子を潰して、その結果コンパートメントがどうなるのか、というのを解析しないといけません。ということは、ターゲットになる遺伝子が一つはあるってことじゃないですか。わたしの場合は、それすらない状態なんです。
マウスの遺伝子って2万個くらいあるんですけど、一つずつ遺伝子を潰していって、全部Hi-Cをやって……ということもできるといえばできるのですが、お金がかかりすぎるんです……。
なるほど。予算も有限ですからね。
そうなんです。ということで、Hi-Cの代わりにDNA複製タイミングを使ってコンパートメント状態を予測しようと考えました。そこでまず、そのDNA複製タイミングを蛍光タンパクを使って簡単に可視化するためのスクリーニング系を立ち上げました。
確立するのに3年がかり
どんなスクリーニングなんですか?
それが、ちょっとややこしいんですけど、大丈夫ですか……。
はい、がんばります(あまり自信ない……)。
私の目的は、A/Bコンパートメントを調節しているタンパク質を探す、です。そのタンパク質を探してこようと思ったら、無作為なスクリーニングが必要になります。
CRISPRという技術があるんですが、遺伝子ノックアウトといって、狙った遺伝子を潰すことができます。この技術を使って、1細胞につき1遺伝子ノックアウトした細胞というのをマウスの全遺伝子(2万種類)分作ります。
ほ、ほう……。
後で、その細胞の配列を読んであげると、どの遺伝子がノックアウトされていたか、というのがわかります。
はい。ここまではわかります。
それと並行して、コンパートメントを調べたい領域に、GFPという蛍光タンパク質を仕込んでおきます。GFPが早く複製されると細胞が強く光って、その領域は複製が早い段階で起きている、ということがわかります。複製が早いということは、先程の相関関係で言うと、その領域はAコンパートメントであるということになります。
で、FACSという装置は、特定の蛍光を発している細胞を選り分けることができるので、GFPが強く光ったものとそうでないものを選別することができる。簡単にいうとそんなスクリーニング系です。
うーん。なるほど…。すごく簡単にいうと、遺伝子を無作為に潰してやって、光ったやつと光らないやつを見てやることで、コンパートメントや複製タイミングに変化があるか?ってのを検出してやる、ってことですかね……?
大体そんな感じです(苦笑)。
でも、これができるまでに、3年かかりました。
3年!
実際、スクリーニング系そのものは、比較的早くできたんですけど、実際にこの系が複製タイミングの変化をきちんと検出できているのか、という実証が必要だったんです。
で、今5年目だから、ようやく結果がで始めた、ってところですかね。
そうですね。可能性がありそうなものが500遺伝子くらい。そのうち今は20遺伝子くらいに絞りました。
さらに、これをコンパートメントが本当に変わっているのかというのを、今から調べないといけないというところです。
道のり長いな!
めっちゃ長いです(笑)。
自由に研究させてもらって
そもそもなんでこんなことをやろうと思ったんですか?
けしてこんなことは初めから思っていたわけではなくて……(笑)。
自分はCRISPRとES細胞Embryonic stem cell(胚性幹細胞)。受精卵が成長した胚から取る。iPSと同様に多能性細胞でさまざまな実験に利用される。が得意だったのもあったので、ES細胞で、核内構造を制御している因子とかをスクリーニングしたら面白そうだなと思って、こういうのをやりませんか?と言ったのがこのラボでの初めのプロジェクトです。
もともと構造に興味があったってことですか?
そうですね。DNA構造に興味を持ちはじめたのは、精子の核内ではDNAが普通の細胞に比べて非常に凝縮されているというのを知ったときですね。ゲノムDNAの巻きつけ方とかもぐちゃぐちゃなわけではもちろんなくて、すごいきれいに巻きつけられてるわけですよ。めっちゃきれいにぐるぐるっと。あんな小さい核の中でそんなことをやっているのがすごい不思議、神秘だって思ったんですよね。
どうしてあんなにキレイに巻けてるのか謎ですよね。
誰か、何かやってるんだろう、と思うんですよね(笑)。
誰かって……。
ちなみに、今やっているスクリーニングは、CRISPRのスクリーニング開発をした京大のラボで一緒にやらせてもらっているんですけど、レンチウイルス細胞に目的の遺伝子を導入する場合に使われる技術。増殖しないよう加工されているが、基準が厳しく設定されており、扱える施設は限られる。を感染させた細胞を使ってFACSするというのが、規定の関係で理研ではできなかったんです。
え?理研でやってないんですか…?
実は、平谷研にももともとはHi-Cをやりたくて来たんですけど、さっきお話ししたように結局Hi-Cはやらなくなってしまって……。
あれ?じゃあ、平谷研じゃなくてもよくないですか……(汗)
結果として、メインのスクリーニングは平谷研の外で行うことになってしまいましたけど、今の研究はひとつの研究室で完結することが少なくなってきていますよね。技術だったり装置だったりを持ち寄って、より良い研究を効率的に進めていくという方向ですね。平谷研では、自由にいろいろとアイデアを試させてもらっているので、ありがたいですね。それにもちろん、これからはスクリーニングで得た遺伝子の変異細胞を使ってHi-Cしていくことになるので、平谷研の強みを活かせると思います!
確かに、いろんな方にお話を伺っていて、BDRの研究はコラボレーションが多い感じがしますね。
あと、外国人研究者も多いし、英語で議論するので、英語に抵抗感がなくなりました(笑)。
じゃあ、次は海外ですか。
そうですね。それも視野に入ってきましたね。
でもとりあえずは、今のスクリーニングの論文を書かないと!
楽しみにしています!

編集後記
いやいや、難しかったです……(汗)。スクリーニングのお話は、実は1度では理解できず、2度3度とお聞きしたものをかなり圧縮しています……。それにしても、一つの結果を出すために、ものすごい地道な作業の積み上げをしているんだと改めて感服。水面下での地道な作業に光を当てるこういう話を書かせてもらっているというのもありがたいことだな、と改めて。
